mRNA vaccine-elicited antibodies to SARS-CoV-2 and circulating variants Zijun Wang et al.
Publicado en la revista Nature https://doi.org/10.1038/s41586-021-03324-6
Resumen
Las vacunas actualmente en uso se basan en provocar inmunidad frente la proteína S, la que forma la espícula, que sirve al virus para reconocer la superficie de la célula humana a la que infecta. Integrada por 1273 aminoácidos, la propteñina S se escinde en dos subunidades (S1 y S2) por proteólisis con furina en los aminoácidos 685-686.
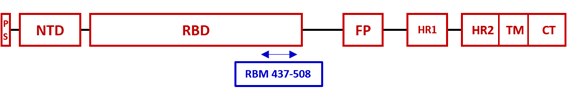
El esquema siguiente ilustra las regiones fundamentales de la proteína. Destaca la región RBD, de reconocimiento del receptor celular y dentro de ella la parte RBM con los motivos fundamentales para esa fijación. En el esquema aparece el péptido-señal, el extremo amino-terminal, el caboxi-terminal y algunos otros motivos característicos. Las distintas mutaciones puntuales que se vienen manejando se designan con el número del aminoácido que cambia por mutación, precedido del que aparece en el virus original y seguido del que lo sustituye por mutación. Por ejemplo, la mutación E484Q implica que el ácido glutámico (E) de la posición 484 ha sido sustituido por glutamina (Q).
- Esta investigación estudia la respuesta inmunitaria en una cohorte de 20 voluntarios, que fueron vacunados con las vacunas de ácido ribonucleico, es decir los productos de Moderna (mRNA-1273) o Pfzer–BioNTech (BNT162b2) frente al SARS-CoV-2.
- Los investigadores manejan plasma de los vacunados con el conjunto de los anticuerpos inducidos, pero también derivan anticuerpos monoclonales a partir de sus células B, para estudiar la especificidad de muchos de los anticuerpos inducidos por la vacunación, incluyendo los lugares de reconocimiento (epítopos) de la proteína por parte de dichos anticuerpos. Igualmente, enfrentan a los sueros policlonales y a los anticuerpos monoclonales con virus portadores de algunas de las mutaciones propias de las variantes de SARS-CoV-2 que se han venido identificando.
- Tras ocho semanas de la segunda dosis, los voluntarios vacunados mostraban elevados niveles de IgM e IgG anti-proteína S y, en concreto, de estos anticuerpos dirigidos contra la región RBD de esta proteína. Estas vacunas de ácido ribonucleico determinan, por tanto, respuestas de anticuerpos frente a la región RBD de manera similar a como lo hace la infección natural, generando anticuerpos que impactan en los mismos epítopos.
- Se puede concluir de todo ello que las vacunas deben provocar la generación de las formas triméricas de la proteína S en el organismo vacunado. Además, la actividad neutralizante del virus y la presencia de células B de memoria en plasma de vacunados resultó equivalente a la encontrada en el plasma de pacientes recuperados de covid contraída por infección natural.
- Sin embargo, la actividad de estos sueros frente a variantes de SARS-CoV-2, portadoras de las mutaciones E484K-, N501Y- ó K417N/E484K/N501 se redujo en proporción limitada pero significativa. Los anticuerpos monoclonales que se inducen por vacunación neutralizan con intensidad al virus SARS-CoV-2 y afectan a diferentes epítopos de la región RBD, lo mismo que ocurre con los monoclonales aislados a partir de los infectados de manera natural. Sin embargo, la capacidad neutralizante de 14 de los anticuerpos monoclonales más potentes se redujo o se perdió totalmente frente a mutantes portadores de los cambios genéticos K417N, E484K o N501Y.
- Los monoclonales humanos neutralizantes que generaron se clasificaron en cuatro categorías, en función de los epítopos RBD sobre los que actúan; las clases 1 y 2 estaban entre los más potentes y abundantes e impactaban en epítopos asociados a los residuos K417, E484 and N501, de ahí la sensibilidad a la selección en función de mutaciones K417N, E484K and N501Y. Para completar sus observaciones, los investigadores ampliaron los estudios construyendo virus recombinantes de SARS-CoV-2 con el virus de la estomatitis vesicular (VSV). Al enfrentar estos virus recombinantes con los anticuerpos monoclonales, inducidos por vacunación, pudieron seleccionar virus mutantes portadores de las referidas mutaciones, por su resistencia a determinados anticuerpos.
- Otras investigaciones publicadas han descrito resultados con plasma de vacunados e infectados, enfrentando a pseudovirus (virus construidos con cada una de las mutaciones) y virus auténticos. Los efectos de reducción de sensibilidad a anticuerpos policlonales son modestos lo que refleja la especificidad múltiple.
- El trabajo indica por tanto que la emergencia de estas variantes en diversos lugares ha tenido lugar por selección inmunitaria. Muy probablemente, dicha selección se produce en infectados con deficiencia inmunitaria con afectación prolongada.
- Concluyen los investigadores que los anticuerpos monoclonales en uso clínico deberían testarse frente a las nuevas variantes. Así mismo, cabe señalar que, aunque la inmunidad policlonal que inducen las vacunas cubre muchas especificidades, la reducción de eficacia que determinan algunas variantes, puede hacer necesario en futuro reformular las vacunas, para cubrir las mutaciones que portan muchas de estas variantes. La tecnología de producción de vacunas (mRNA vírico o adenovirus vector), basadas en la información genética para la proteína S, facilita esta reformulación de las vacunas si fura necesaria.
 Newsletter
Newsletter Contacto
Contacto




